Главная > Новости >
В фармацевтических исследованиях часто малые молекулы ("фрагменты") тестируются на эффективное связывание со специфическими белковыми мишенями. Из-за малого размера, фрагменты могут химически выделяться и связываться друг с другом, в итоге давая желаемые структуры с высокой аффинностью[1]. (Рис. 1).
Новый Frag Xtal Screen основан на подходе "Сначала кристаллография": рентгентовская кристаллография показывает не только будут ли фрагменты связываться с белком, но также где и как будет происходить связывание. Кристаллы исследуемых белков замачиваются с отдельными фрагментами и производится прямой сбор их структурных данных.
Новый Frag Xtal Screen...
... состоит из 96 фрагментов, покрывающих большой химический диапазон, подобранные на основе работы групп Gerhard Klebe (University of Marburg) и Manfred Weiss (BESSY II)
...избегает потери потенциальных попаданий, не идентифицированных методами предварительной проверки [3,4]
Предполагая наличие практических навыков в переносе кристаллов и криозаморозке, 96 отдельных фрагментов могут быть проверены в течение одной недели.
* эффективность = наибольшая энергия связывания/молекулярную массу
Источники:
[1] Rees et al. (2004) Fragment-based lead discovery Nat. Rev. Drug Discov. 3:660.
[2] Huschmann et al. (2016) Structures of endothiapepsin-fragment complexes from crystallographic fragment screening using a novel, diverse and affordable 96-compound fragment library. Acta Cryst F 72:346.
[3] Schiebel et al. (2016) Six Biophysical Screening Methods Miss a Large Proportion of Crystallographic Discovered Fragment Hits: A Case Study. ACS Chem. Biol. 11:1693.
[4] Schiebel et al. (2015) One Question, Multiple Answers: Biochemical and Biophysical Screening Methods Retrieve Deviating Fragment Hit Lists. ChemMedChem 10:1511.
[5] www.ebi.ac.uk/pdbe-srv/pdbechem
Фрагментный скрининг стал проще: Frag Xtal Screen
10.02.2017
В фармацевтических исследованиях часто малые молекулы ("фрагменты") тестируются на эффективное связывание со специфическими белковыми мишенями. Из-за малого размера, фрагменты могут химически выделяться и связываться друг с другом, в итоге давая желаемые структуры с высокой аффинностью[1]. (Рис. 1).
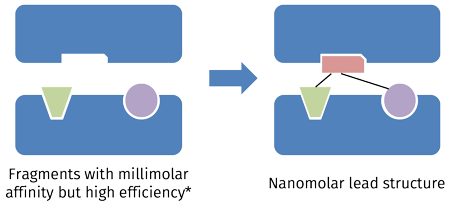
| Рис.1. Основанное на фрагментах исследование использует преимущество эволюции и связывания фрагментов. Малые отдельные фрагменты с низкой собственной аффинностью, но высокой эффективностью* растут в соответствии со структурной моделью. В результате наблюдается эффективное связывание фрагментов в наномолярной структуре [1]. |
Новый Frag Xtal Screen основан на подходе "Сначала кристаллография": рентгентовская кристаллография показывает не только будут ли фрагменты связываться с белком, но также где и как будет происходить связывание. Кристаллы исследуемых белков замачиваются с отдельными фрагментами и производится прямой сбор их структурных данных.
Новый Frag Xtal Screen...
... состоит из 96 фрагментов, покрывающих большой химический диапазон, подобранные на основе работы групп Gerhard Klebe (University of Marburg) и Manfred Weiss (BESSY II)
- априорная проверка через выбор соединений в записях PDBeChem [5]
- доказанная способность фрагментов связываться с белками [2,5]
- проверка с помощью аспаргиновой протеазы (эндотиапепсин, EP), 10% попаданий [2]
...позволяет замачивать кристаллы с фрагментами в концентрации > 90 mM, что приводит к функциональному картированию мишени целиком и идентификации множественных сайтов связывания.
- доказанная способность фрагментов связываться с белками [2,5]
- проверка с помощью аспаргиновой протеазы (эндотиапепсин, EP), 10% попаданий [2]
...избегает потери потенциальных попаданий, не идентифицированных методами предварительной проверки [3,4]
Предполагая наличие практических навыков в переносе кристаллов и криозаморозке, 96 отдельных фрагментов могут быть проверены в течение одной недели.
* эффективность = наибольшая энергия связывания/молекулярную массу
Источники:
[1] Rees et al. (2004) Fragment-based lead discovery Nat. Rev. Drug Discov. 3:660.
[2] Huschmann et al. (2016) Structures of endothiapepsin-fragment complexes from crystallographic fragment screening using a novel, diverse and affordable 96-compound fragment library. Acta Cryst F 72:346.
[3] Schiebel et al. (2016) Six Biophysical Screening Methods Miss a Large Proportion of Crystallographic Discovered Fragment Hits: A Case Study. ACS Chem. Biol. 11:1693.
[4] Schiebel et al. (2015) One Question, Multiple Answers: Biochemical and Biophysical Screening Methods Retrieve Deviating Fragment Hit Lists. ChemMedChem 10:1511.
[5] www.ebi.ac.uk/pdbe-srv/pdbechem